- Home
- La voce della comunità
- Leggere il DNA per combattere le malattie genetiche

Leggere il DNA per combattere le malattie genetiche
| Maddalena Vario | La voce della comunità
All'Istituto Telethon, grazie ad avanzate tecnologie di sequenziamento del genoma, si identificano i geni responsabili delle mutazioni genetiche
Uno dei progetti in corso al TIGEM ha l’obiettivo di leggere il DNA per evidenziare le mutazioni genetiche responsabili delle malattie genetiche. In particolare attraverso la tecnologia del Next Generation Sequencing (NGS) si arriva prima a capire quali basi del DNA dei pazienti affetti da malattie genetiche sono mutate rispetto alle basi del DNA di persone sane e poi a identificare il gene che è responsabile della malattia.
Ne abbiamo parlato con l’ingegner Diego di Bernardo, direttore del Bioinformatics Core del TIGEM, l’Istituto Telethon di Genetica e Medicina di Napoli.
 Diego Di Bernardo
Diego Di Bernardo
TIGEM, Telethon Institute of Genetics and Medicine
Direttore Programma di ricerca in biologia dei sistemi e genomica funzionale
Ingegner di Bernardo, in cosa consiste la tecnica del Next Generation Sequencing per leggere il DNA?
Il nostro approccio allo studio delle malattie genetiche fa sì che le stesse vengano viste di fatto come un circuito in cui si è rotto un componente. La tecnica del NGS non solo offre affidabilità diagnostica di gran lunga maggiore rispetto alle tecniche precedenti, ma soprattutto rende possibile l’analisi mediante sequenziamento di regioni molto vaste del genoma. Con la NGS è possibile effettuare un’indagine genetica senza dover necessariamente ipotizzare un gene responsabile a priori, ma sequenziare un pannello molto ampio di geni o, per finalità di ricerca, tutto il genoma. In particolare il gene può essere visto come uno dei componenti di un complesso motore (la cellula) e quindi, per riparare una cellula malata, occorre prima individuare quale parte del motore ha il problema.
TIGEM: ISTITUTO TELETHON DI GENETICA E MEDICINA

Fondato nel '94, il TIGEM ha sede a Pozzuoli (NA). La sua mission è centrata sulla comprensione dei meccanismi alla base delle malattie genetiche, per poi sviluppare strategie preventive e terapeutiche. Il lavoro di ricerca segue tre programmi strategici: biologia cellulare delle malattie genetiche, biologia dei sistemi e della genomica funzionale e terapia molecolare. Connesso in banda ultralarga alla rete GARR in fibra ottica.
Grazie a questo approccio di straordinaria potenza, possiamo analizzare grandi quantitativi di DNA in tempi relativamente rapidi con notevoli risparmi. Con il nostro strumento per NGS che permette di sequenziare, cioè leggere il genoma, possiamo effettuare questo screening su 30 milioni di basi, che compongono i 25.000 geni, per decine di pazienti in un singolo esperimento. Il genoma, infatti, contiene 3 miliardi di basi ma solo l’1% fanno parte dei geni, il 99% delle rimanenti basi contiene sequenze la cui funzione è ancora in fase di studio.
Evidenziare le mutazioni genetiche responsabili della malattia, potrà aiutare i clinici negli ospedali nella diagnosi e quindi nella cura?
Al momento l’uso delle nostre analisi è solo per i ricercatori e non per i clinici negli ospedali, ma l’idea è fare in modo che in un futuro prossimo la classificazione delle mutazioni che individuiamo possa guidare i clinici che devono poi interpretare i dati. Classificare le mutazioni è importante se si considera che in ogni individuo possono essere individuate più di 20.000 mutazioni rispetto alla popolazione di riferimento e da queste mutazioni si deve poi arrivare a capire qual è quella causa della malattia.
Avete anche la possibilità di accedere a banche dati?
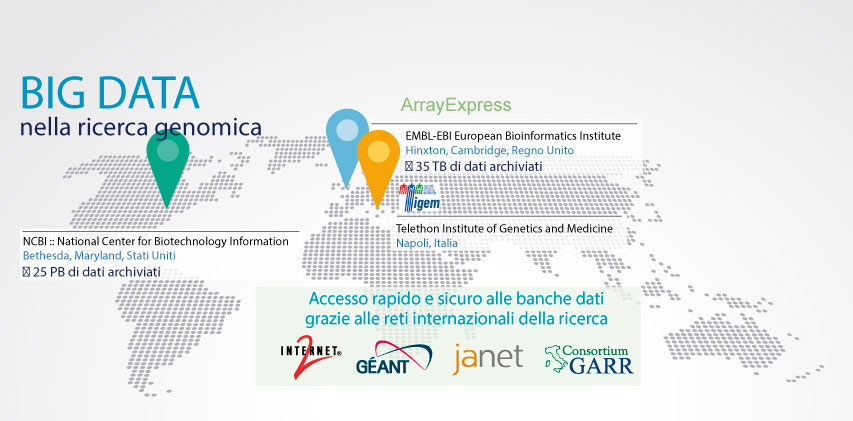
Certo. I dati che vengono prodotti nei nostri laboratori, così come quelli di tutte le Università ed Istituti di ricerca nel mondo sono depositati in banche dati accessibili da tutti, come ArrayExpress, presso EMBL-EBI (European Bioinformatics Institute) e Gene Expression Omnibus (GEO), presso NCBI (National Center for Biotechnology Information), collegate alle rispettive reti della ricerca nazionali (Janet e Internet2), per cui essere sulla rete della ricerca GARR, interconnessa alle altre attraverso la rete europea GÉANT, permette di accedervi in maniera immediata e sicura. Queste banche dati contengono oltre che dati da cellule di pazienti, anche dati da linee cellulari, ovvero cellule che crescono in cultura o perché cellule tumorali o perché immortalizzate cioè modificate geneticamente e quindi utilizzate nei laboratori di tutto il mondo per fare esperimenti. Si tratta di cellule che si riproducono come dei batteri, si congelano e si possono distribuire per essere analizzate.
In questo caso, attraverso tecniche sperimentali si può rimuovere da queste cellule il gene responsabile della malattia nei pazienti, e si può così osservare il comportamento della cellula per capire perché quel gene, quando non funziona, causa la malattia genetica. Avere a disposizione i dati genomici di queste cellule nelle banche dati è una grande ricchezza per i ricercatori perché permette di poter effettuare delle analisi ad ampio spettro. Sempre al TIGEM stiamo infatti seguendo un progetto molto interessante sul riposizionamento dei farmaci per la terapia delle malattie genetiche rare.
Riposizionare i farmaci partendo dall’analisi delle cellule. Interessante, in che modo ciò avviene?
Mi spiego meglio. Grazie alle banche dati, possiamo prelevare tutti i dati sperimentali che ci indicano in che modo una linea cellulare risponde ad un determinato farmaco. Ci sono migliaia di esperimenti di questo tipo per migliaia di farmaci. Inoltre, possiamo anche prelevare i dati sperimentali delle linee cellulari in cui un gene-malattia è stato rimosso. Incrociando questi dati è possibile identificare quei farmaci che potrebbero avere un effetto terapeutico per alcune malattie genetiche. Ad esempio è noto da secoli che l’aspirina (il cui componente attivo è l’acido salicilico originariamente estratto dal salice piangente) ha un effetto antinfiammatorio ma non si sapeva quale fosse il meccanismo molecolare alla base di questa sua azione, scoperto solo recentemente.
 Sempre in relazione all’aspirina, sono stati scoperti solo recentemente i suoi effetti di fluidificazione del sangue, la cui consapevolezza fa in modo che oggi venga utilizzato dai cardiopatici (cardio-aspirina). Sono state scoperte fatte per caso, ebbene quello che noi facciamo è razionalizzare questi processi, ricercando un modo sistematico di analizzare questi dati (ad esempio tramite strumenti di Machine Learning e statistica avanzata). E proprio nell’ambito delle malattie rare di cui ci occupiamo, di riposizionamento ce n’è davvero bisogno, dato che le case farmaceutiche poco investono nella ricerca in questo campo: immagini dunque come può diventare preziosa l’informazione che un farmaco già in uso possa essere utilizzato anche per la cura di malattie genetiche rare. Una volta identificato un metodo di analisi, la nostra ricerca viene poi condivisa in pubblicazioni.
Sempre in relazione all’aspirina, sono stati scoperti solo recentemente i suoi effetti di fluidificazione del sangue, la cui consapevolezza fa in modo che oggi venga utilizzato dai cardiopatici (cardio-aspirina). Sono state scoperte fatte per caso, ebbene quello che noi facciamo è razionalizzare questi processi, ricercando un modo sistematico di analizzare questi dati (ad esempio tramite strumenti di Machine Learning e statistica avanzata). E proprio nell’ambito delle malattie rare di cui ci occupiamo, di riposizionamento ce n’è davvero bisogno, dato che le case farmaceutiche poco investono nella ricerca in questo campo: immagini dunque come può diventare preziosa l’informazione che un farmaco già in uso possa essere utilizzato anche per la cura di malattie genetiche rare. Una volta identificato un metodo di analisi, la nostra ricerca viene poi condivisa in pubblicazioni.
Di quali macchine disponete per effettuare queste analisi?
Si tratta di cluster di computer per il calcolo parallelo, dato che possono essere prodotti fino a un terabyte di dati per esperimento che poi devono essere analizzati. Al momento le nostre macchine sono in locale, in quanto il numero di dati sperimentali presenti nelle banche dati è ancora tale da permetterci di analizzare un numero gestibile di dati. Vorrei precisare che solo grazie alla rete GARR siamo in grado di avere accesso e prelevare terabyte di dati dalle banche dati internazionali in un tempo ragionevole. Inoltre, grazie al miglioramento delle tecnologie NGS, i dati prodotti dai laboratori nazionali ed internazionali sono destinati a crescere esponenzialmente, in futuro anche l’analisi di questi dati avverrà non più in locale ma in remoto con il cloud computing per cui avere accesso alla rete GARR sarà fondamentale per poter fare ricerca
 Il genoma umano
Il genoma umano
Il genoma umano è una lunga catena di circa tre miliardi di basi (A, C, G e T) che compongono il DNA. Nel genoma sono stati identificati circa 25.000 geni. Ogni gene è una sequenze di migliaia di basi che codificano gli elementi funzionali di una cellula (proteine e RNA non-codificanti).
IL GRUPPO DI RICERCA DI NEXT GENERATION SEQUENCING
Il responsabile genetista del progetto Next Generation Sequencing (NGS) Facility del TIGEM è Vincenzo Nigro, professore universitario alla Seconda Università di Napoli. Il gruppo di ricerca da lui diretto si occupa di leggere il genoma delle cellule, composto di miliardi di “lettere” chiamate basi, e trasformarle in dati gestibili da un calcolatore. Questi dati vengono poi analizzati dal Bioinformatics Core del TIGEM diretto dall’Ingegner Diego di Bernardo, Direttore del programma di ricerca in biologia dei sistemi e genomica funzionale del TIGEM e ricercatore alla Università Federico II di Napoli.
www.tigem.it
Dai un voto da 1 a 5, ne terremo conto per scrivere i prossimi articoli.
Voto attuale:
Ultimi articoli in rubrica
-
di Marta Mieli
-
di Ufficio Stampa ENEA
-
di Ufficio Stampa INGV
-
di Matteo Massicci
-
di Mario Pireddu, Stefano Moriggi
 Telegram
Telegram